
基础组分
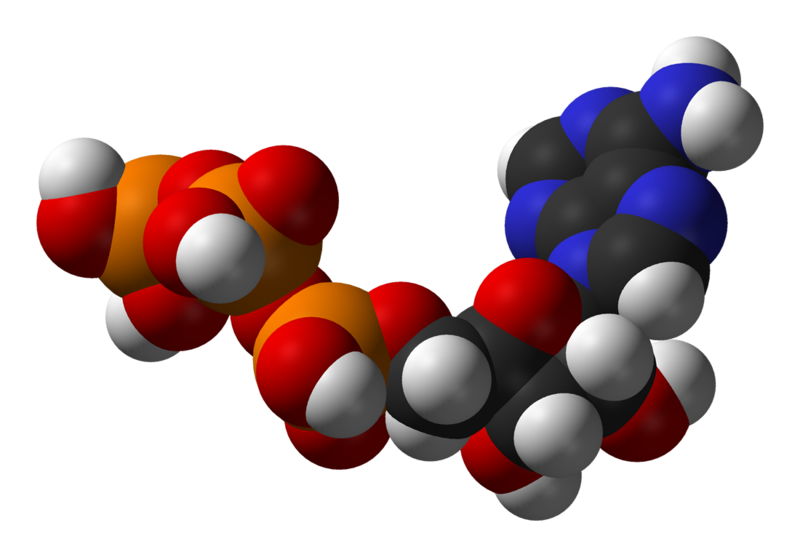
三磷酸腺苷(ATP)的结构,是能量代谢中的中间体
代谢(/mətæbəlɪzəm/,来自希腊语:μεταβολήde代谢,“改变”)是生物体内维持生命的化学反应的集合。新陈代谢的三个主要目的是将食物/燃料转化为能量以进行细胞过程,将食物/燃料转化为蛋白质,脂质,核酸和一些碳水化合物的构建块,以及消除含氮废物。这些酶催化反应使生物体能够生长和繁殖,维持其结构并对其环境做出反应。新陈代谢这个词也可以指生物体内发生的所有化学反应的总和,包括消化和物质进入不同细胞和在不同细胞之间的运输,在这种情况下,细胞内的一组反应被称为中间代谢或中间代谢。
代谢通常分为两类:分解代谢,例如有机物的分解,葡萄糖分解为丙酮酸,细胞呼吸和合成代谢,蛋白质和核酸等细胞成分的积累。通常,分解释放能量和积累消耗能量。
代谢的化学反应被组织成代谢途径,其中一种化学物质通过一系列步骤通过一系列步骤转化为另一种化学物质。酶对新陈代谢至关重要,因为它们可以让生物体通过将它们与释放能量的自发反应联系起来,从而产生需要自身不会发生的能量的理想反应。酶充当催化剂,使反应更快地进行。酶还允许调节代谢途径以响应细胞环境的变化或来自其他细胞的信号。
视频:↓ 能量食物代谢动画
https://cache.tv.qq.com/qqplayerout.swf?vid=k07870pukms
特定生物体的代谢系统决定了哪些物质会发现营养丰富,哪些有毒。例如,一些原核生物使用硫化氢作为营养物质,但这种气体对动物有毒。新陈代谢的速度,代谢率,影响生物体需要多少食物,也影响它如何获得食物。
新陈代谢的一个显著特征是即使是截然不同的物种之间的基本代谢途径和组分的相似性。例如,在柠檬酸循环中最熟知的一组羧酸存在于所有已知的生物体中,存在于多种细菌如大肠杆菌和巨大的多细胞生物如大象中。代谢途径中这些惊人的相似之处可能是由于它们在进化史上的早期出现,以及它们因其功效而保留。
目录
1 关键生化药剂
1.1 氨基酸和蛋白质
1.2 脂质
1.3 碳水化合物
1.4 核苷酸
1.5 辅酶
1.6 矿物和辅助因子
2 分解代谢
2.1 消化
2.2 有机化合物的能量
3 能量转换
3.1 氧化磷酸化
3.2 无机化合物的能量
3.3 来自光的能量
4 合成代谢
4.1 碳固定
4.2 碳水化合物和聚糖
4.3 脂肪酸,类异戊二烯和类固醇
4.4 蛋白质
4.5 核苷酸合成和补救
5 异生素和氧化还原代谢
6 生物体的热力学
7 调节和控制
8 进化
9 调查和操纵
10 参考文献
关键生化药剂
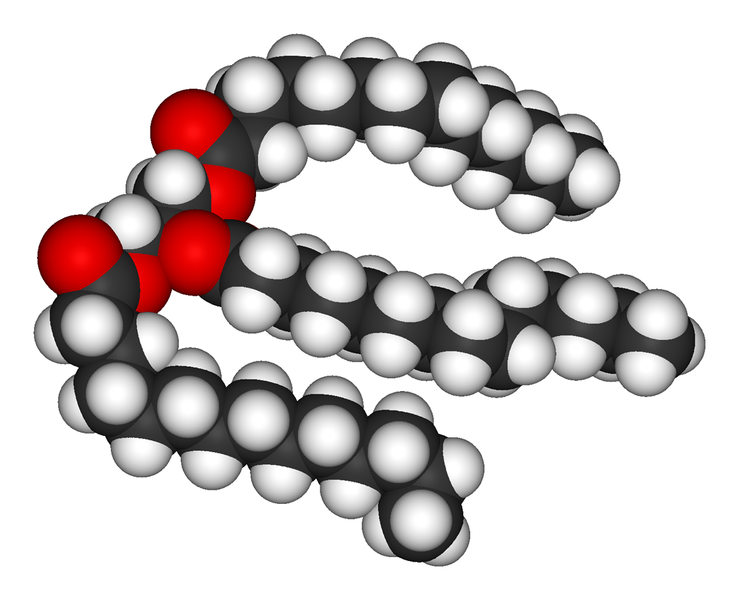
三酰基甘油脂质的结构
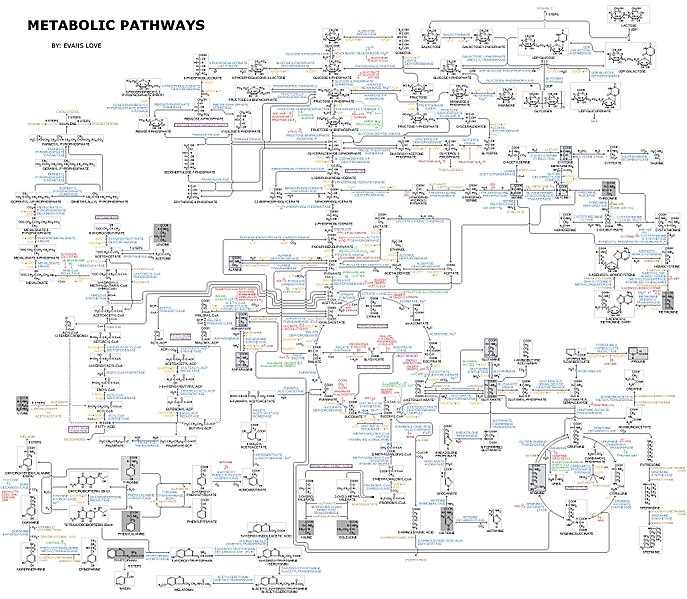
这是描绘大量人代谢途径的图。
构成动物,植物和微生物的大多数结构由三种基本类别的分子组成:氨基酸,碳水化合物和脂质(通常称为脂肪)。由于这些分子对生命至关重要,因此代谢反应或者集中在制造细胞和组织的过程中制造这些分子,或者通过消化将它们分解并用作能量来源。这些生物化学物质可以连接在一起,形成聚合物,如DNA和蛋白质,生命必需的大分子。
氨基酸和蛋白质
蛋白质由排列成通过肽键连接在一起的直链中的氨基酸组成。许多蛋白质是催化代谢中化学反应的酶。其他蛋白质具有结构或机械功能,例如形成细胞骨架的功能,即维持细胞形状的支架系统。蛋白质在细胞信号传导,免疫应答,细胞粘附,跨膜的主动转运和细胞周期中也很重要。氨基酸还通过提供进入柠檬酸循环(三羧酸循环)的碳源而促进细胞能量代谢,特别是当主要能量源如葡萄糖缺乏时,或当细胞经历代谢应激时。
血脂
脂质是最多样化的生化组。它们的主要结构用途是作为内部和外部生物膜的一部分,例如细胞膜,或作为能量来源。脂质通常被定义为疏水性或两亲性生物分子,但会溶解在有机溶剂中,例如苯或氯仿。脂肪是一大类含有脂肪酸和甘油的化合物;与三种脂肪酸酯连接的甘油分子称为三酰基甘油酯。存在这种基本结构的若干变体,包括交替的主链,例如鞘脂中的鞘氨醇,和磷脂中的亲水基团,例如磷酸盐。类固醇如胆固醇是另一类主要的脂质。
碳水化合物
直链形式由连续连接的四个CHOH基团组成,末端被醛基COH和甲醇基团CH 2 O H封端。为了形成环,醛基与下一个的OH基团结合。 - 在甲醇组之前的另一端的碳。
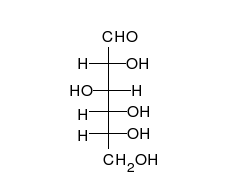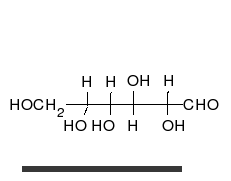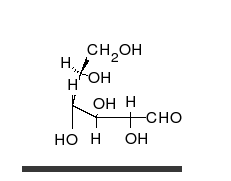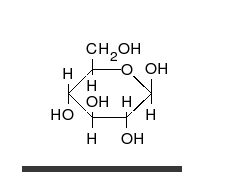
葡萄糖可以以直链形式和环形存在。
碳水化合物是醛或酮,附着有许多羟基,可以直链或环的形式存在。碳水化合物是最丰富的生物分子,并且充当许多角色,例如能量(淀粉,糖原)和结构组分(植物中的纤维素,动物中的几丁质)的储存和运输。基本碳水化合物单元称为单糖,包括半乳糖,果糖,最重要的是葡萄糖。单糖可以以几乎无限的方式连接在一起形成多糖。
核苷酸
两种核酸DNA和RNA是核苷酸的聚合物。每个核苷酸由连接到核糖或脱氧核糖基团上的磷酸盐组成,其附着于含氮碱基。核酸对于遗传信息的存储和使用以及通过转录和蛋白质生物合成过程的解释至关重要。该信息受DNA修复机制保护,并通过DNA复制传播。许多病毒具有RNA基因组,例如HIV,其使用逆转录从其病毒RNA基因组创建DNA模板。核酶如剪接体和核糖体中的RNA与酶类似,因为它可以催化化学反应。通过将核碱基连接至核糖来制备单个核苷。这些碱是含有氮的杂环,分类为嘌呤或嘧啶。核苷酸在代谢组转移反应中也充当辅酶。
辅酶
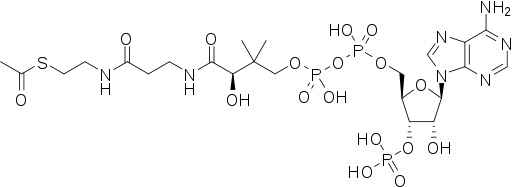
辅酶乙酰-CoA的结构。可转移的乙酰基与最左侧的硫原子键合。
代谢涉及大量的化学反应,但大多数都属于一些基本类型的反应,这些反应涉及原子的官能团及其在分子内的键的转移。这种常见的化学特性允许细胞使用一小组代谢中间体在不同反应之间携带化学基团。这些基团转移中间体称为辅酶。每类基团转移反应都是由一种特殊的辅酶进行的,这种辅酶是产生它的一组酶的底物,也是一组消耗它的酶。因此,这些辅酶不断地制造,消耗然后再循环。
一种中心辅酶是三磷酸腺苷(ATP),它是细胞的通用能量货币。该核苷酸用于在不同的化学反应之间转移化学能。细胞中只有少量的ATP,但随着它不断再生,人体每天可以使用自身的ATP重量。 ATP是分解代谢和合成代谢之间的桥梁。分解代谢分解分子,合成代谢将它们组合在一起。分解代谢反应产生ATP,合成代谢反应消耗它。它还在磷酸化反应中充当磷酸基团的载体。
维生素是一种无法在细胞中制造的少量有机化合物。在人体营养中,大多数维生素在修饰后起辅酶的作用;例如,当它们用于细胞时,所有水溶性维生素都被磷酸化或与核苷酸偶联。烟酰胺腺嘌呤二核苷酸(NAD +)是维生素B3(烟酸)的衍生物,是一种重要的辅酶,可作为氢受体。数百种不同类型的脱氢酶从其底物中去除电子并将NAD +还原成NADH。然后,这种还原形式的辅酶是细胞中需要还原其底物的任何还原酶的底物。烟酰胺腺嘌呤二核苷酸在细胞中以两种相关形式存在,NADH和NADPH。 NAD + / NADH形式在分解代谢反应中更重要,而NADP + / NADPH用于合成代谢反应。
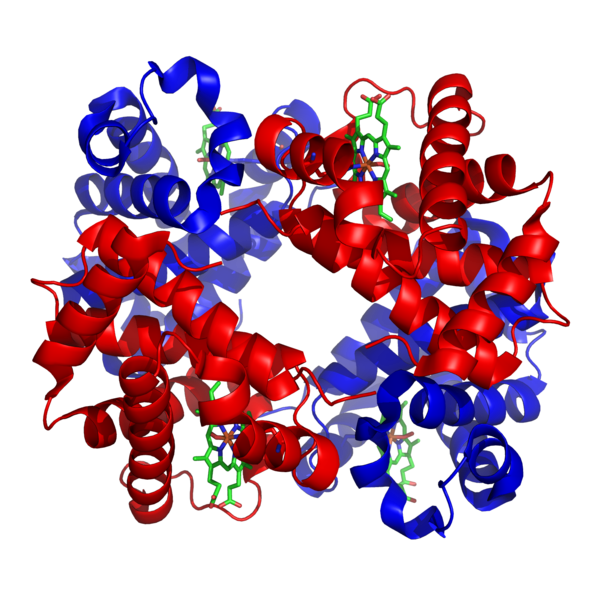
血红蛋白的结构。蛋白质亚基为红色和蓝色,含铁血红素基团为绿色。来自PDB:1GZX。
矿物质和辅助因子
无机元素在新陈代谢中起着关键作用;一些是丰富的(例如钠和钾),而另一些则在微小浓度下起作用。约99%的哺乳动物体重由碳,氮,钙,钠,氯,钾,氢,磷,氧和硫元素组成。有机化合物(蛋白质,脂类和碳水化合物)含有大部分碳和氮;大部分氧气和氢气以水的形式存在。
丰富的无机元素充当离子电解质。最重要的离子是钠,钾,钙,镁,氯化物,磷酸盐和有机离子碳酸氢盐。跨细胞膜维持精确的离子梯度可维持渗透压和pH值。离子对于神经和肌肉功能也是至关重要的,因为这些组织中的动作电位是通过细胞外液和细胞液(胞质溶胶)之间的电解质交换产生的。电解质通过称为离子通道的细胞膜中的蛋白质进入和离开细胞。例如,肌肉收缩取决于钙,钠和钾通过细胞膜和T-管中的离子通道的运动。
过渡金属通常作为微量元素存在于生物体中,其中锌和铁是最丰富的。这些金属在一些蛋白质中用作辅助因子,并且对于诸如过氧化氢酶和氧载体蛋白质如血红蛋白的酶的活性是必需的。金属辅助因子与蛋白质中的特定位点紧密结合;尽管酶辅因子可以在催化过程中被修饰,但它们总是在催化反应结束时恢复到其原始状态。金属微量营养素通过特定转运蛋白吸收到生物体中,并在不使用时与储存蛋白质如铁蛋白或金属硫蛋白结合。
分解代谢
分解代谢是分解大分子的一组代谢过程。这些包括分解和氧化食物分子。分解代谢反应的目的是提供构成分子的合成代谢反应所需的能量和组分。这些分解代谢反应的确切性质因生物体而异,生物体可根据其能量来源和碳(其主要营养成分)进行分类,如下表所示。有机分子被有机营养物用作能量来源,而岩藻营养物使用无机底物,而光养生物则将太阳光捕获为化学能。然而,所有这些不同形式的代谢依赖于氧化还原反应,其涉及电子从还原的供体分子(例如有机分子,水,氨,硫化氢或亚铁离子)转移到受体分子(例如氧,硝酸盐或硫酸盐)。在动物中,这些反应涉及复杂的有机分子,这些分子被分解成更简单的分子,例如二氧化碳和水。在光合生物中,例如植物和蓝细菌,这些电子转移反应不释放能量,而是用作储存从太阳光吸收的能量的方式。
动物中最常见的分解代谢反应可分为三个主要阶段。在第一阶段,大的有机分子,如蛋白质,多糖或脂质,被消化到细胞外的较小组分中。接下来,这些较小的分子被细胞吸收并转化为较小的分子,通常是乙酰辅酶A(乙酰辅酶A),释放出一些能量。最后,CoA上的乙酰基在柠檬酸循环和电子传递链中被氧化成水和二氧化碳,释放通过将辅酶烟酰胺腺嘌呤二核苷酸(NAD +)还原成NADH而储存的能量。
消化
淀粉,纤维素或蛋白质等大分子不能被细胞迅速吸收,必须先将其分解成较小的单位才能用于细胞代谢。 几种常见的酶消化这些聚合物。 这些消化酶包括将蛋白质消化成氨基酸的蛋白酶,以及将多糖消化成称为单糖的单糖的糖苷水解酶。
微生物只是将消化酶分泌到周围环境中,而动物只能从肠内的特化细胞中分泌这些酶,包括胃和胰腺,以及唾液腺。 然后通过这些细胞外酶释放的氨基酸或糖通过活性转运蛋白泵入细胞。

蛋白质,碳水化合物和脂肪分解代谢的简化概述
有机化合物的能量
碳水化合物分解代谢是碳水化合物分解成更小的单位。碳水化合物一旦被消化成单糖,通常会被带入细胞。进入内部后,主要的分解途径是糖酵解,其中糖类如葡萄糖和果糖转化为丙酮酸,并产生一些ATP。丙酮酸是几种代谢途径中的中间体,但大多数通过需氧(含氧)糖酵解转化为乙酰辅酶A并进入柠檬酸循环。尽管在柠檬酸循环中产生了更多的ATP,但最重要的产物是NADH,其在乙酰-CoA被氧化时由NAD +制成。该氧化作为废物释放二氧化碳。在厌氧条件下,糖酵解产生乳酸,通过酶乳酸脱氢酶将NADH再氧化成NAD +以再次用于糖酵解。葡萄糖分解的另一种途径是戊糖磷酸途径,其减少辅酶NADPH并产生戊糖,例如核酸,核酸的糖组分。
通过水解将脂肪分解成游离脂肪酸和甘油。甘油进入糖酵解,脂肪酸被β氧化分解,释放乙酰辅酶A,然后进入柠檬酸循环。脂肪酸在氧化时比碳水化合物释放更多的能量,因为碳水化合物在其结构中含有更多的氧。在类似于β氧化的过程中,类固醇也被一些细菌分解,并且这种分解过程涉及释放大量的乙酰-CoA,丙酰-CoA和丙酮酸,它们都可以被细胞用于能量。结核分枝杆菌也可以作为碳的唯一来源在脂质胆固醇上生长,并且在结核分枝杆菌的感染生命周期的各个阶段中已经证实参与胆固醇使用途径的基因是重要的。
氨基酸或者用于合成蛋白质和其他生物分子,或者被氧化成尿素和二氧化碳作为能量来源。氧化途径始于转氨酶去除氨基。将氨基进料到尿素循环中,留下酮酸形式的脱氨碳骨架。这些酮酸中的几种是柠檬酸循环中的中间体,例如谷氨酸的脱氨基形成α-酮戊二酸。还可以通过糖异生(下面讨论)将葡萄糖原氨基酸转化为葡萄糖。
能量转换
氧化磷酸化
在氧化磷酸化中,在诸如原酸循环的区域中从有机分子中去除的电子被转移到氧气中,并且释放的能量用于产生ATP。 这是在真核生物中通过称为电子传递链的线粒体膜中的一系列蛋白质完成的。 在原核生物中,这些蛋白质存在于细胞的内膜中。 这些蛋白质利用从NADH等还原分子传递电子释放到氧气上的能量,将质子泵送到膜上。
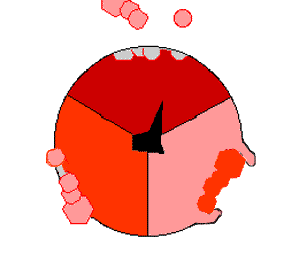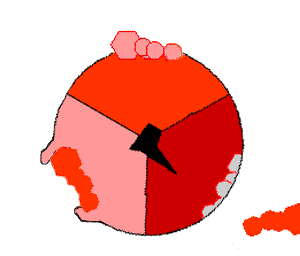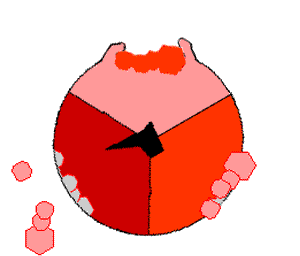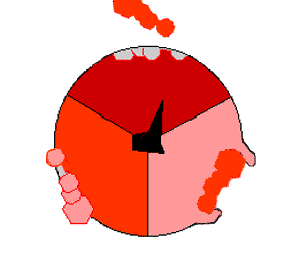
ATP合成酶的作用机制。 ATP显示为红色,ADP和磷酸盐显示为粉红色,旋转茎状亚基显示为黑色。
将质子泵出线粒体会在膜上产生质子浓度差异并产生电化学梯度。这种力通过称为ATP合酶的酶的基础将质子驱回线粒体。质子的流动使茎亚基旋转,导致合酶结构域的活性位点改变形状并磷酸化二磷酸腺苷 - 将其转化为ATP。
无机化合物的能量
化合营养是在原核生物中发现的一种代谢,其中能量来自无机化合物的氧化。这些生物可以使用氢,还原硫化合物(如硫化物,硫化氢和硫代硫酸盐),亚铁(FeII)或氨作为还原能源,它们通过电子受体如氧气或亚硝酸盐氧化这些化合物获得能量。这些微生物过程在全球生物地球化学循环中很重要,如乙酸生成,硝化和反硝化,对土壤肥力至关重要。
来自光的能量
阳光中的能量被植物,蓝细菌,紫色细菌,绿色硫细菌和一些原生生物捕获。该过程通常与二氧化碳转化为有机化合物相结合,作为光合作用的一部分,这将在下面讨论。然而,能量捕获和碳固定系统可以在原核生物中单独操作,因为紫色细菌和绿色硫细菌可以使用阳光作为能量来源,同时在碳固定和有机化合物的发酵之间切换。
在许多生物体中,太阳能的捕获原则上类似于氧化磷酸化,因为它涉及将能量存储为质子浓度梯度。然后,这种质子动力驱动ATP合成。驱动这种电子传递链所需的电子来自称为光合作用反应中心或视紫红质的聚光蛋白。根据存在的光合色素的类型,反应中心分为两类,大多数光合细菌只有一种类型,而植物和蓝细菌有两种。
在植物,藻类和蓝细菌中,光系统II使用光能从水中去除电子,释放氧气作为废物。然后电子流向细胞色素b6f复合物,它利用它们的能量将质子泵送到叶绿体中的类囊体膜上。像以前一样,这些质子在驱动ATP合酶时会向后移动通过膜。然后电子流过光系统I,然后可以用于还原辅酶NADP +,用于下面讨论的卡尔文循环,或者再循环用于进一步产生ATP。
合成代谢
合成代谢是一组建设性代谢过程,其中分解代谢释放的能量用于合成复杂分子。通常,构成细胞结构的复杂分子是从小而简单的前体逐步构建的。合成代谢涉及三个基本阶段。首先,生产氨基酸,单糖,类异戊二烯和核苷酸等前体,其次,利用ATP的能量将其活化为活性形式,第三,将这些前体组装成复杂的分子,如蛋白质,多糖,脂类和核酸。
生物体根据细胞中构建的分子数量而不同。 植物等自养生物可以在细胞中构建复杂的有机分子,如多糖和来自二氧化碳和水等简单分子的蛋白质。 另一方面,异养生物需要更复杂物质的来源,例如单糖和氨基酸,以产生这些复杂分子。 生物可以通过其能量的最终来源进一步分类:光合自养生物和光合异养生物从光获得能量,而化学自养生物和化学异养生物从无机氧化反应获得能量。
碳固定
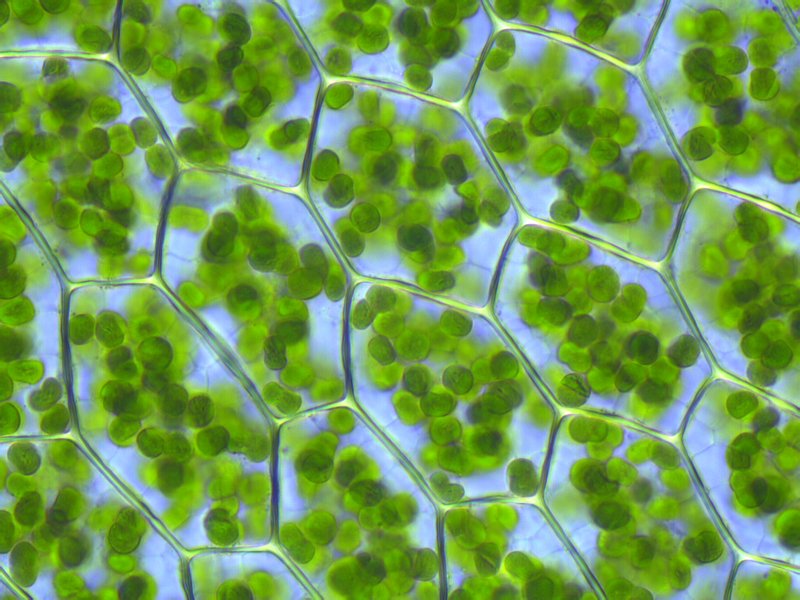
植物细胞(以紫色墙为界)充满叶绿体(绿色),这是光合作用的部位
光合作用是来自太阳光和二氧化碳(CO2)的碳水化合物的合成。在植物,蓝细菌和藻类中,含氧光合作用分解水,氧气作为废物产生。该过程使用由光合反应中心产生的ATP和NADPH,如上所述,将CO 2转化为甘油酸3-磷酸,然后可将其转化为葡萄糖。这种碳固定反应由RuBisCO酶进行,作为Calvin-Benson循环的一部分。植物中发生三种光合作用,C3碳固定,C4碳固定和CAM光合作用。这些不同之处在于二氧化碳进入卡尔文循环的途径,C3工厂直接固定CO2,而C4和CAM光合作用首先将CO2结合到其他化合物中,以适应强烈的阳光和干燥条件。
在光合原核生物中,碳固定的机制更加多样化。在这里,二氧化碳可以通过Calvin - Benson循环,反向柠檬酸循环或乙酰辅酶A的羧化来固定。原核化学自养生物也通过Calvin - Benson循环固定CO2,但使用无机化合物的能量来驱动反应。
碳水化合物和聚糖
在碳水化合物合成代谢中,简单的有机酸可以转化为单糖如葡萄糖,然后用于组装多糖如淀粉。从丙酮酸盐,乳酸盐,甘油,甘油酸3-磷酸和氨基酸等化合物产生葡萄糖称为糖异生。糖原异生通过一系列中间体将丙酮酸转化为葡萄糖-6-磷酸,其中许多中间体与糖酵解共享。然而,该途径不仅仅是糖酵解反向运行,因为几个步骤由非糖酵解酶催化。这很重要,因为它可以分别调节葡萄糖的形成和分解,并防止两种途径在无效循环中同时运行。
虽然脂肪是储存能量的常用方式,但在人类等脊椎动物中,这些储存中的脂肪酸不能通过糖异生转化为葡萄糖,因为这些生物不能将乙酰辅酶A转化为丙酮酸;植物,但动物没有,有必要的酶机制。因此,在长期饥饿后,脊椎动物需要从脂肪酸中生产酮体,以取代不能代谢脂肪酸的大脑等组织中的葡萄糖。在植物和细菌等其他生物体中,这种代谢问题通过乙醛酸循环解决,该循环绕过柠檬酸循环中的脱羧步骤并允许乙酰辅酶A转化为草酰乙酸,其中它可用于生产葡萄糖。
多糖和聚糖是通过糖基转移酶从反应性糖 - 磷酸供体如尿苷二磷酸葡萄糖(UDP-葡萄糖)依次加入单糖到生长多糖上的受体羟基而制备的。 由于基质环上的任何羟基可以是受体,所产生的多糖可具有直链或支链结构。 产生的多糖本身可具有结构或代谢功能,或通过称为寡糖基转移酶的酶转移至脂质和蛋白质。
脂肪酸,类异戊二烯和类固醇
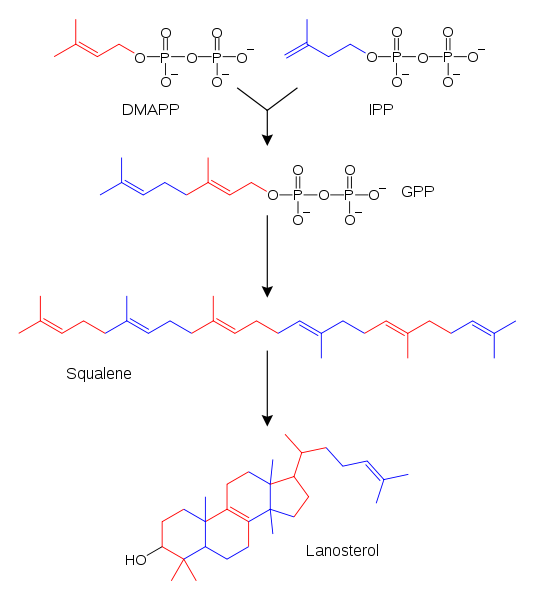
具有中间体异戊烯焦磷酸(IPP),二甲基烯丙基焦磷酸(DMAPP),香叶基焦磷酸(GPP)和角鲨烯的类固醇合成途径的简化形式显示。为清楚起见,省略了一些中间体。
脂肪酸由脂肪酸合成酶制成,其聚合然后还原乙酰-CoA单元。脂肪酸中的酰基链通过一系列反应延长,所述反应添加酰基,将其还原为醇,将其脱水为烯烃基团,然后再将其还原为烷烃基团。脂肪酸生物合成的酶分为两组:在动物和真菌中,所有这些脂肪酸合酶反应都是通过单一的多功能I型蛋白质进行的,而在植物质体和细菌中,分离的II型酶在通路中的每一步都进行。
萜烯和类异戊二烯是一大类脂类,包括类胡萝卜素,并形成最大类的植物天然产物。这些化合物是通过组装和改性由反应性前体异戊烯焦磷酸和二甲基烯丙基焦磷酸盐提供的异戊二烯单元制备的。这些前体可以以不同方式制造。在动物和古细菌中,甲羟戊酸途径从乙酰辅酶A产生这些化合物,而在植物和细菌中,非甲羟戊酸途径使用丙酮酸和3-磷酸甘油醛作为底物。使用这些活化的异戊二烯供体的一个重要反应是类固醇生物合成。这里,异戊二烯单元连接在一起制成角鲨烯,然后折叠起来形成一组环以制备羊毛甾醇。然后可以将羊毛甾醇转化为其他类固醇,例如胆固醇和麦角甾醇。
蛋白质
生物体合成20种常见氨基酸的能力各不相同。大多数细菌和植物可合成二十种,但哺乳动物只能合成十一种非必需氨基酸,因此必须从食物中获得九种必需氨基酸。一些简单的寄生虫,如肺炎支原体(Mycoplasma pneumoniae),缺乏所有氨基酸合成,并直接从宿主中摄取氨基酸。所有氨基酸均由糖酵解,柠檬酸循环或戊糖磷酸途径中的中间体合成。氮由谷氨酸和谷氨酰胺提供。氨基酸合成取决于合适的α-酮酸的形成,然后将其转氨基形成氨基酸。
氨基酸通过在肽键链中连接在一起而制成蛋白质。每种不同的蛋白质都有独特的氨基酸残基序列:这是它的主要结构。就像字母表中的字母可以组合形成几乎无穷无尽的单词一样,氨基酸可以以不同的顺序连接,形成多种多样的蛋白质。蛋白质由氨基酸制成,所述氨基酸通过酯键与转移RNA分子连接而被激活。该氨酰基-tRNA前体在由氨酰基tRNA合成酶进行的ATP依赖性反应中产生。然后该氨酰基-tRNA是核糖体的底物,其使用信使RNA中的序列信息将氨基酸连接到延伸的蛋白质链上。
核苷酸合成和回收
核苷酸由需要大量代谢能的途径中的氨基酸,二氧化碳和甲酸制成。因此,大多数生物体具有有效的系统来挽救预先形成的核苷酸。嘌呤合成为核苷(与核糖连接的碱基)。腺嘌呤和鸟嘌呤均由前体核苷肌苷一磷酸制成,其使用来自氨基酸甘氨酸,谷氨酰胺和天冬氨酸的原子以及从辅酶四氢叶酸转移的甲酸盐合成。另一方面,嘧啶类由碱性乳清酸合成,其由谷氨酰胺和天冬氨酸形成。
异生素和氧化还原代谢
所有生物都经常暴露于它们不能用作食物的化合物,如果它们在细胞中积累则会有害,因为它们没有代谢功能。这些具有潜在破坏性的化合物被称为异生素。异生素如合成药物,天然毒物和抗生素通过一系列异生物质代谢酶解毒。在人类中,这些包括细胞色素P450氧化酶,UDP-葡糖醛酸基转移酶,和谷胱甘肽S-转移酶。该酶系统在三个阶段起作用,首先氧化异生物质(阶段I),然后将水溶性基团缀合到分子上(阶段II)。然后可以将修饰的水溶性异生物质泵出细胞,并且在多细胞生物体中可以在排泄之前进一步代谢(阶段III)。在生态学中,这些反应在污染物的微生物生物降解和受污染的土地和石油泄漏的生物修复中尤为重要。许多这些微生物反应与多细胞生物共享,但由于微生物类型的难以置信的多样性,这些生物能够处理比多细胞生物更广泛的异生物,并且甚至可以降解持久性有机污染物,例如有机氯化物。
需氧生物的相关问题是氧化应激。这里,包括氧化磷酸化和在蛋白质折叠过程中形成二硫键的过程产生活性氧物质,例如过氧化氢。这些有害的氧化剂被抗氧化剂代谢物如谷胱甘肽和酶如过氧化氢酶和过氧化物酶除去。
生物体的热力学
生物体必须遵守热力学定律,这些定律描述了热量和功的传递。热力学第二定律指出,在任何封闭系统中,熵(无序)的数量都不会减少。尽管生物体的惊人复杂性似乎与这一法律相矛盾,但生命是可能的,因为所有生物体都是与周围环境交换物质和能量的开放系统。因此,生命系统不是平衡的,而是耗散系统,通过引起环境熵的更大增加来维持其高复杂性状态。细胞的新陈代谢通过将分解代谢的自发过程与合成代谢的非自发过程联系起来实现。在热力学方面,新陈代谢通过创造障碍维持秩序。
调控
由于大多数生物的环境不断变化,因此必须对新陈代谢的反应进行精细调节,以在细胞内维持一组恒定的条件,这种情况称为体内平衡。代谢调节还允许有机体响应信号并与其环境积极地相互作用。两个密切相关的概念对于理解代谢途径如何被控制非常重要。首先,通路中酶的调节是其响应信号的活性增加和减少的方式。其次,该酶发挥的控制作用是其活性的这些变化对通路的总体速率(通过通路的通量)的影响。例如,酶可以显示出大的活性变化(即它受到高度调节),但如果这些变化对代谢途径的通量几乎没有影响,则该酶不参与该途径的控制。
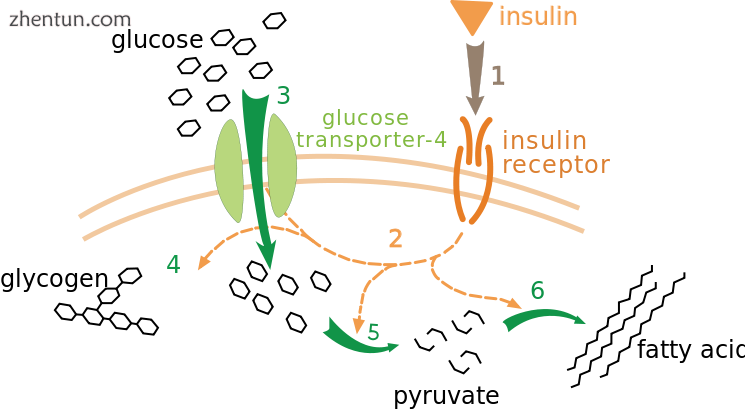
胰岛素对葡萄糖摄取和代谢的影响。胰岛素与其受体(1)结合,后者又启动许多蛋白激活级联反应(2)。这些包括:Glut-4转运蛋白易位至质膜和葡萄糖流入(3),糖原合成(4),糖酵解(5)和脂肪酸合成(6)。
有多种水平的代谢调节。在内在调节中,代谢途径自我调节以响应底物或产物水平的变化;例如,产品量的减少可以增加通过通道的通量以进行补偿。这种类型的调节通常涉及对途径中多种酶的活性的变构调节。外在控制涉及多细胞生物体中的细胞,其响应于来自其他细胞的信号而改变其代谢。这些信号通常以可溶性信使的形式存在,例如激素和生长因子,并且由细胞表面上的特定受体检测。然后这些信号通过第二信使系统在细胞内传播,第二信使系统通常涉及蛋白质的磷酸化。
一个非常好理解的外在控制的例子是激素胰岛素对葡萄糖代谢的调节。胰岛素是响应血糖水平升高而产生的。激素与细胞上的胰岛素受体的结合然后激活一系列蛋白激酶,其导致细胞摄取葡萄糖并将其转化为储存分子,例如脂肪酸和糖原。糖原的代谢受磷酸化酶的活性控制,磷酸化酶是分解糖原的酶,而糖原合成酶是制造糖原的酶。这些酶以相互的方式调节,磷酸化抑制糖原合酶,但激活磷酸化酶。胰岛素通过激活蛋白磷酸酶并导致这些酶的磷酸化降低而导致糖原合成。
演化
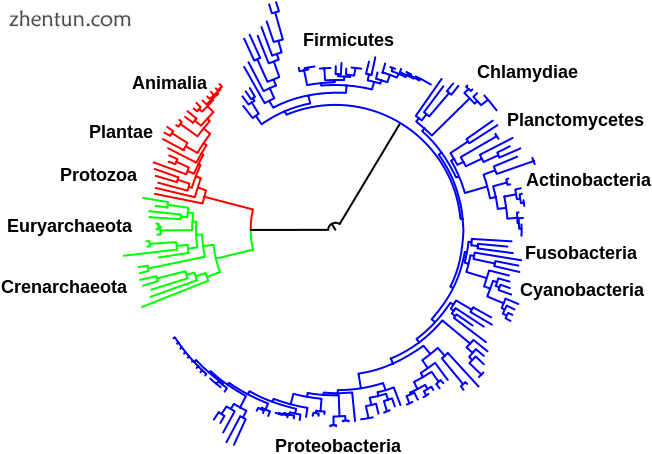
进化树显示来自生命的所有三个领域的生物的共同祖先。细菌呈蓝色,真核生物呈红色,古细菌呈绿色。包含的一些门的相对位置显示在树周围。
上述代谢的中心途径,例如糖酵解和柠檬酸循环,存在于生物的所有三个领域中,并存在于最后的普遍共同祖先中。这种通用的祖先细胞是原核细胞,可能是具有广泛氨基酸,核苷酸,碳水化合物和脂质代谢的产甲烷菌。在后来的进化过程中保留这些古老的途径可能是这些反应已成为其特定代谢问题的最佳解决方案的结果,其中诸如糖酵解和柠檬酸循环的途径高效且以最少的步骤产生其最终产物。基于酶的代谢的第一途径可能是嘌呤核苷酸代谢的一部分,而先前的代谢途径是古代RNA世界的一部分。
已经提出许多模型来描述新的代谢途径进化的机制。这些包括顺序添加新的酶到短的祖先途径,整个途径的重复和分歧以及预先存在的酶的募集和它们组装成新的反应途径。这些机制的相对重要性尚不清楚,但基因组研究表明,一条途径中的酶可能具有共同的祖先,这表明许多途径已经逐步发展,并且具有从先前存在的步骤创建的新功能。在路上。另一种模型来自追踪代谢网络中蛋白质结构演变的研究,这表明酶是普遍招募的,借用酶在不同的代谢途径中执行类似的功能(在MANET数据库中很明显)这些招募过程导致进化酶促马赛克。第三种可能性是代谢的某些部分可能作为“模块”存在,其可以在不同途径中重复使用并且在不同分子上执行类似的功能。
除了新的代谢途径的进化,进化也可以导致代谢功能的丧失。例如,在一些寄生虫中,丧失对生存不重要的代谢过程,并且可以替代地从宿主中清除预先形成的氨基酸,核苷酸和碳水化合物。在内共生生物中也观察到类似的降低的代谢能力。
调查和操纵
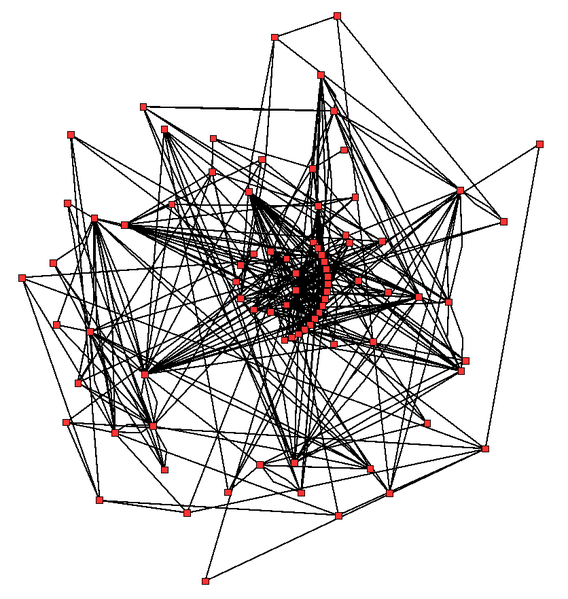
拟南芥柠檬酸循环的代谢网络。酶和代谢物显示为红色方块,它们之间的相互作用为黑色线条。
传统上,通过简化方法研究新陈代谢,该方法侧重于单一代谢途径。特别有价值的是在整个生物体,组织和细胞水平上使用放射性示踪剂,其通过鉴定放射性标记的中间体和产物来定义从前体到最终产物的途径。然后可以纯化催化这些化学反应的酶,并研究它们的动力学和对抑制剂的反应。平行方法是识别细胞或组织中的小分子;这些分子的完整组合称为代谢组。总体而言,这些研究可以很好地了解简单代谢途径的结构和功能,但是当应用于更复杂的系统(例如完整细胞的代谢)时,这些研究是不充分的。
图中给出了含有数千种不同酶的细胞中代谢网络复杂性的概念,该图显示了右侧仅43种蛋白质和40种代谢物之间的相互作用:基因组序列提供了含有多达45,000种基因的列表。然而,现在可以使用这种基因组数据重建完整的生化反应网络,并产生更全面的数学模型,可以解释和预测它们的行为。当用于将通过经典方法获得的途径和代谢物数据与来自蛋白质组学和DNA微阵列研究的基因表达数据整合时,这些模型特别有用。使用这些技术,现在已经产生了人体代谢模型,其将指导未来的药物发现和生物化学研究。这些模型现在用于网络分析,将人类疾病分为具有共同蛋白质或代谢物的群体。
细菌代谢网络是蝴蝶结组织的一个突出例子,这种结构能够输入多种营养素,并使用相对较少的中间普通货币生产各种各样的产品和复杂的大分子。
该信息的主要技术应用是代谢工程。 在这里,对酵母,植物或细菌等生物进行遗传修饰,使其在生物技术中更有用,并有助于生产抗生素或工业化学品如1,3-丙二醇和莽草酸等药物。 这些遗传修饰通常旨在减少用于生产产品的能量,增加产量并减少废物的产生。
历史
新陈代谢这个术语来源于希腊语Μεταβολισμός - “Metabolismos”用于“改变”或“推翻”。
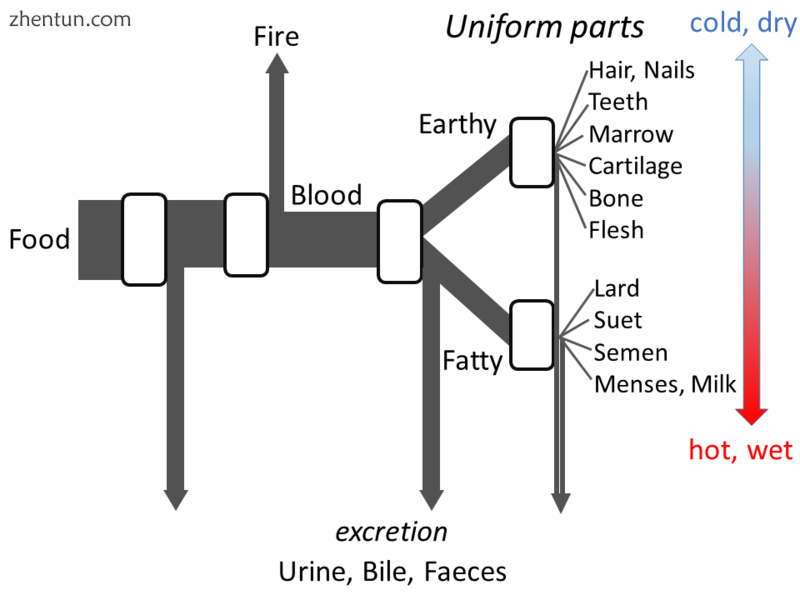
亚里士多德的新陈代谢作为开放流动模型
亚里士多德的“动物的部分”详细阐述了他对新陈代谢的看法,以制定开放式流动模型。他认为,在这个过程的每个阶段,食物中的物质都会被转化,热量作为经典的火元素释放出来,剩余的物质会以尿液,胆汁或粪便的形式排出体外。
Ibn al-Nafis描述了公元1260年作品中的新陈代谢,名为Al-Risalah al-Kamiliyyah fil Siera al-Nabawiyyah(先知传记中卡米尔的论文),其中包括以下短语“身体及其部位都处于连续状态解散和营养,所以它们不可避免地会发生永久性变化。“新陈代谢科学研究的历史跨越了几个世纪,并且从早期研究中检查整个动物,到检查现代生物化学中的个体代谢反应。人类新陈代谢的第一个对照实验由Santorio Santorio于1614年在其著作“Ars de statica medicina”中出版。他描述了他在吃饭,睡觉,工作,性生活,禁食,饮酒和排泄之前和之后的体重。他发现他摄入的大部分食物都是通过他所谓的“无知的汗水”而丢失的。

Santorio Santorio在他的杆秤平衡中,来自Ars de statica medicina,于1614年首次出版
在这些早期研究中,尚未发现这些代谢过程的机制,并且认为生命组织具有生命力。在19世纪,当研究通过酵母将糖发酵成酒精时,路易斯巴斯德得出结论,发酵是由酵母细胞内的物质催化的,他称之为“酵素”。他写道,“酒精发酵是一种与酵母细胞的生命和组织相关的行为,而不是细胞的死亡或腐败。”这一发现,以及FriedrichWöhler于1828年发表的一篇关于尿素化学合成的论文,以及作为第一种由全无机前体制备的有机化合物而闻名。这证明细胞中发现的有机化合物和化学反应在原理上与化学的任何其他部分没有区别。
Eduard Buchner在20世纪初发现了酶,它将代谢的化学反应研究与细胞的生物学研究分开,并标志着生物化学的开始。生物化学知识的大量在20世纪初迅速增长。汉斯克雷布斯是这些现代生物化学家中最多产的人之一,他为新陈代谢研究做出了巨大贡献。他发现尿素循环后来与Hans Kornberg一起研究柠檬酸循环和乙醛酸循环。现代生物化学研究得到了新技术的发展,如色谱,X射线衍射,核磁共振光谱,放射性同位素标记,电子显微镜和分子动力学模拟。这些技术允许发现和详细分析细胞中的许多分子和代谢途径。
另见
icon Metabolism portal
Underwater diving portal
Anthropogenic metabolism
Antimetabolite
Basal metabolic rate
Calorimetry
Isothermal microcalorimetry
Inborn error of metabolism
Iron-sulfur world theory, a "metabolism first" theory of the origin of life
Metabolic disorder
Primary nutritional groups
Respirometry
Stream metabolism
Sulfur metabolism
Thermic effect of food
Urban metabolism
Water metabolism
Overflow metabolism
Reactome
KEGG
参考
Friedrich C (1998). "Physiology and genetics of sulfur-oxidizing bacteria". Adv Microb Physiol. Advances in Microbial Physiology. 39: 235–89. doi:10.1016/S0065-2911(08)60018-1. ISBN 978-0-12-027739-1. PMID 9328649.
Pace NR (January 2001). "The universal nature of biochemistry". Proc. Natl. Acad. Sci. U.S.A. 98 (3): 805–8. Bibcode:2001PNAS...98..805P. doi:10.1073/pnas.98.3.805. PMC 33372. PMID 11158550.
Smith E, Morowitz H (2004). "Universality in intermediary metabolism". Proc Natl Acad Sci USA. 101 (36): 13168–73. Bibcode:2004PNAS..10113168S. doi:10.1073/pnas.0404922101. PMC 516543. PMID 15340153.
Ebenhöh O, Heinrich R (2001). "Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems". Bull Math Biol. 63 (1): 21–55. doi:10.1006/bulm.2000.0197. PMID 11146883.
Meléndez-Hevia E, Waddell T, Cascante M (1996). "The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution". J Mol Evol. 43 (3): 293–303. Bibcode:1996JMolE..43..293M. doi:10.1007/BF02338838. PMID 8703096.
Michie K, Löwe J (2006). "Dynamic filaments of the bacterial cytoskeleton". Annu Rev Biochem. 75: 467–92. doi:10.1146/annurev.biochem.75.103004.142452. PMID 16756499.
Nelson, David L.; Michael M. Cox (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company. p. 841. ISBN 0-7167-4339-6.
Kelleher J, Bryan 3rd, B, Mallet R, Holleran A, Murphy A, and Fiskum G (1987). "Analysis of tricarboxylic acid-cycle metabolism of hepatoma cells by comparison of 14CO2 ratios". Biochem J. 246 (3): 633–639. doi:10.1042/bj2460633. PMC 1148327. PMID 3120698.
Hothersall, J & Ahmed, A (2013). "Metabolic fate of the increased yeast amino acid uptake subsequent to catabolite derepression". J Amino Acids. 2013: e461901. doi:10.1155/2013/461901. PMC 3575661. PMID 23431419.
Fahy E, Subramaniam S, Brown H, Glass C, Merrill A, Murphy R, Raetz C, Russell D, Seyama Y, Shaw W, Shimizu T, Spener F, van Meer G, VanNieuwenhze M, White S, Witztum J, Dennis E (2005). "A comprehensive classification system for lipids". J Lipid Res. 46 (5): 839–61. doi:10.1194/jlr.E400004-JLR200. PMID 15722563.
"Nomenclature of Lipids". IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Retrieved 8 March 2007.
Hegardt F (1999). "Mitochondrial 3-hydroxy-3-methylglutaryl-CoA synthase: a control enzyme in ketogenesis". Biochem J. 338 (Pt 3): 569–82. doi:10.1042/0264-6021:3380569. PMC 1220089. PMID 10051425.
Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R (2005). "Glycomics: an integrated systems approach to structure-function relationships of glycans". Nat Methods. 2 (11): 817–24. doi:10.1038/nmeth807. PMID 16278650.
Sierra S, Kupfer B, Kaiser R (2005). "Basics of the virology of HIV-1 and its replication". J Clin Virol. 34 (4): 233–44. doi:10.1016/j.jcv.2005.09.004. PMID 16198625.
Wimmer M, Rose I (1978). "Mechanisms of enzyme-catalyzed group transfer reactions". Annu Rev Biochem. 47: 1031–78. doi:10.1146/annurev.bi.47.070178.005123. PMID 354490.
Mitchell P (1979). "The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems". Eur J Biochem. 95 (1): 1–20. doi:10.1111/j.1432-1033.1979.tb12934.x. PMID 378655.
Dimroth P, von Ballmoos C, Meier T (March 2006). "Catalytic and mechanical cycles in F-ATP synthases: Fourth in the Cycles Review Series". EMBO Rep. 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397.
Coulston, Ann; Kerner, John; Hattner, JoAnn; Srivastava, Ashini (2006). "Nutrition Principles and Clinical Nutrition". Stanford School of Medicine Nutrition Courses. SUMMIT.
Pollak N, Dölle C, Ziegler M (2007). "The power to reduce: pyridine nucleotides – small molecules with a multitude of functions". Biochem J. 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440. PMID 17295611.
Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R (1991). "Chemical and elemental analysis of humans in vivo using improved body composition models". Am J Physiol. 261 (2 Pt 1): E190–8. PMID 1872381.
Sychrová H (2004). "Yeast as a model organism to study transport and homeostasis of alkali metal cations" (PDF). Physiol Res. 53 Suppl 1: S91–8. PMID 15119939.
Levitan I (1988). "Modulation of ion channels in neurons and other cells". Annu Rev Neurosci. 11: 119–36. doi:10.1146/annurev.ne.11.030188.001003. PMID 2452594.
Dulhunty A (2006). "Excitation-contraction coupling from the 1950s into the new millennium". Clin Exp Pharmacol Physiol. 33 (9): 763–72. doi:10.1111/j.1440-1681.2006.04441.x. PMID 16922804.
Mahan D, Shields R (1998). "Macro- and micromineral composition of pigs from birth to 145 kilograms of body weight" (PDF). J Anim Sci. 76 (2): 506–12. PMID 9498359.
Husted S, Mikkelsen B, Jensen J, Nielsen N (2004). "Elemental fingerprint analysis of barley (Hordeum vulgare) using inductively coupled plasma mass spectrometry, isotope-ratio mass spectrometry, and multivariate statistics". Anal Bioanal Chem. 378 (1): 171–82. doi:10.1007/s00216-003-2219-0. PMID 14551660.
Finney L, O'Halloran T (2003). "Transition metal speciation in the cell: insights from the chemistry of metal ion receptors". Science. 300 (5621): 931–6. Bibcode:2003Sci...300..931F. doi:10.1126/science.1085049. PMID 12738850.
Cousins R, Liuzzi J, Lichten L (2006). "Mammalian zinc transport, trafficking, and signals". J Biol Chem. 281 (34): 24085–9. doi:10.1074/jbc.R600011200. PMID 16793761.
Dunn L, Rahmanto Y, Richardson D (2007). "Iron uptake and metabolism in the new millennium". Trends Cell Biol. 17 (2): 93–100. doi:10.1016/j.tcb.2006.12.003. PMID 17194590.
Nealson K, Conrad P (1999). "Life: past, present and future". Philos Trans R Soc Lond B Biol Sci. 354 (1392): 1923–39. doi:10.1098/rstb.1999.0532. PMC 1692713. PMID 10670014.
Nelson N, Ben-Shem A (2004). "The complex architecture of oxygenic photosynthesis". Nat Rev Mol Cell Biol. 5 (12): 971–82. doi:10.1038/nrm1525. PMID 15573135.
Häse C, Finkelstein R (December 1993). "Bacterial extracellular zinc-containing metalloproteases". Microbiol Rev. 57 (4): 823–37. PMC 372940. PMID 8302217.
Gupta R, Gupta N, Rathi P (2004). "Bacterial lipases: an overview of production, purification and biochemical properties". Appl Microbiol Biotechnol. 64 (6): 763–81. doi:10.1007/s00253-004-1568-8. PMID 14966663.
Hoyle T (1997). "The digestive system: linking theory and practice". Br J Nurs. 6 (22): 1285–91. PMID 9470654.
Souba W, Pacitti A (1992). "How amino acids get into cells: mechanisms, models, menus, and mediators". JPEN J Parenter Enteral Nutr. 16 (6): 569–78. doi:10.1177/0148607192016006569. PMID 1494216.
Barrett M, Walmsley A, Gould G (1999). "Structure and function of facilitative sugar transporters". Curr Opin Cell Biol. 11 (4): 496–502. doi:10.1016/S0955-0674(99)80072-6. PMID 10449337.
Bell G, Burant C, Takeda J, Gould G (1993). "Structure and function of mammalian facilitative sugar transporters". J Biol Chem. 268 (26): 19161–4. PMID 8366068.
Bouché C, Serdy S, Kahn C, Goldfine A (2004). "The cellular fate of glucose and its relevance in type 2 diabetes". Endocr Rev. 25 (5): 807–30. doi:10.1210/er.2003-0026. PMID 15466941.
Wipperman, Matthew, F.; Thomas, Suzanne, T.; Sampson, Nicole, S. (2014). "Pathogen roid rage: Cholesterol utilization by Mycobacterium tuberculosis". Crit. Rev. Biochem. Mol. Biol. 49 (4): 269–93. doi:10.3109/10409238.2014.895700. PMC 4255906. PMID 24611808.
Sakami W, Harrington H (1963). "Amino acid metabolism". Annu Rev Biochem. 32: 355–98. doi:10.1146/annurev.bi.32.070163.002035. PMID 14144484.
Brosnan J (2000). "Glutamate, at the interface between amino acid and carbohydrate metabolism". J Nutr. 130 (4S Suppl): 988S–90S. PMID 10736367.
Young V, Ajami A (2001). "Glutamine: the emperor or his clothes?". J Nutr. 131 (9 Suppl): 2449S–59S, discussion 2486S–7S. PMID 11533293.
Hosler J, Ferguson-Miller S, Mills D (2006). "Energy Transduction: Proton Transfer Through the Respiratory Complexes". Annu Rev Biochem. 75: 165–87. doi:10.1146/annurev.biochem.75.062003.101730. PMC 2659341. PMID 16756489.
Schultz B, Chan S (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes". Annu Rev Biophys Biomol Struct. 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051.
Capaldi R, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem Sci. 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513.
Friedrich B, Schwartz E (1993). "Molecular biology of hydrogen utilization in aerobic chemolithotrophs". Annu Rev Microbiol. 47: 351–83. doi:10.1146/annurev.mi.47.100193.002031. PMID 8257102.
Weber K, Achenbach L, Coates J (2006). "Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction". Nat Rev Microbiol. 4 (10): 752–64. doi:10.1038/nrmicro1490. PMID 16980937.
Jetten M, Strous M, van de Pas-Schoonen K, Schalk J, van Dongen U, van de Graaf A, Logemann S, Muyzer G, van Loosdrecht M, Kuenen J (1998). "The anaerobic oxidation of ammonium". FEMS Microbiol Rev. 22 (5): 421–37. doi:10.1111/j.1574-6976.1998.tb00379.x. PMID 9990725.
Simon J (2002). "Enzymology and bioenergetics of respiratory nitrite ammonification". FEMS Microbiol Rev. 26 (3): 285–309. doi:10.1111/j.1574-6976.2002.tb00616.x. PMID 12165429.
Conrad R (1996). "Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)". Microbiol Rev. 60 (4): 609–40. PMC 239458. PMID 8987358.
Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005). "Microbial co-operation in the rhizosphere". J Exp Bot. 56 (417): 1761–78. doi:10.1093/jxb/eri197. PMID 15911555.
van der Meer M, Schouten S, Bateson M, Nübel U, Wieland A, Kühl M, de Leeuw J, Sinninghe Damsté J, Ward D (July 2005). "Diel Variations in Carbon Metabolism by Green Nonsulfur-Like Bacteria in Alkaline Siliceous Hot Spring Microbial Mats from Yellowstone National Park". Appl Environ Microbiol. 71 (7): 3978–86. doi:10.1128/AEM.71.7.3978-3986.2005. PMC 1168979. PMID 16000812.
Tichi M, Tabita F (2001). "Interactive Control of Rhodobacter capsulatus Redox-Balancing Systems during Phototrophic Metabolism". J Bacteriol. 183 (21): 6344–54. doi:10.1128/JB.183.21.6344-6354.2001. PMC 100130. PMID 11591679.
Allen J, Williams J (1998). "Photosynthetic reaction centers". FEBS Lett. 438 (1–2): 5–9. doi:10.1016/S0014-5793(98)01245-9. PMID 9821949.
Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (2004). "Cyclic electron flow around photosystem I is essential for photosynthesis". Nature. 429 (6991): 579–82. Bibcode:2004Natur.429..579M. doi:10.1038/nature02598. PMID 15175756.
Miziorko H, Lorimer G (1983). "Ribulose-1,5-bisphosphate carboxylase-oxygenase". Annu Rev Biochem. 52: 507–35. doi:10.1146/annurev.bi.52.070183.002451. PMID 6351728.
Dodd A, Borland A, Haslam R, Griffiths H, Maxwell K (2002). "Crassulacean acid metabolism: plastic, fantastic". J Exp Bot. 53 (369): 569–80. doi:10.1093/jexbot/53.369.569. PMID 11886877.
Hügler M, Wirsen C, Fuchs G, Taylor C, Sievert S (May 2005). "Evidence for Autotrophic CO2 Fixation via the Reductive Tricarboxylic Acid Cycle by Members of the ɛ Subdivision of Proteobacteria". J Bacteriol. 187 (9): 3020–7. doi:10.1128/JB.187.9.3020-3027.2005. PMC 1082812. PMID 15838028.
Strauss G, Fuchs G (1993). "Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle". Eur J Biochem. 215 (3): 633–43. doi:10.1111/j.1432-1033.1993.tb18074.x. PMID 8354269.
Wood H (1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB J. 5 (2): 156–63. PMID 1900793.
Shively J, van Keulen G, Meijer W (1998). "Something from almost nothing: carbon dioxide fixation in chemoautotrophs". Annu Rev Microbiol. 52: 191–230. doi:10.1146/annurev.micro.52.1.191. PMID 9891798.
Boiteux A, Hess B (1981). "Design of glycolysis". Philos Trans R Soc Lond B Biol Sci. 293 (1063): 5–22. Bibcode:1981RSPTB.293....5B. doi:10.1098/rstb.1981.0056. PMID 6115423.
Pilkis S, el-Maghrabi M, Claus T (1990). "Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics". Diabetes Care. 13 (6): 582–99. doi:10.2337/diacare.13.6.582. PMID 2162755.
Ensign S (2006). "Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation". Mol Microbiol. 61 (2): 274–6. doi:10.1111/j.1365-2958.2006.05247.x. PMID 16856935.
Finn P, Dice J (2006). "Proteolytic and lipolytic responses to starvation". Nutrition. 22 (7–8): 830–44. doi:10.1016/j.nut.2006.04.008. PMID 16815497.
Kornberg H, Krebs H (1957). "Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle". Nature. 179 (4568): 988–91. Bibcode:1957Natur.179..988K. doi:10.1038/179988a0. PMID 13430766.
Rademacher T, Parekh R, Dwek R (1988). "Glycobiology". Annu Rev Biochem. 57: 785–838. doi:10.1146/annurev.bi.57.070188.004033. PMID 3052290.
Opdenakker G, Rudd P, Ponting C, Dwek R (1993). "Concepts and principles of glycobiology". FASEB J. 7 (14): 1330–7. PMID 8224606.
McConville M, Menon A (2000). "Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)". Mol Membr Biol. 17 (1): 1–16. doi:10.1080/096876800294443. PMID 10824734.
Chirala S, Wakil S (2004). "Structure and function of animal fatty acid synthase". Lipids. 39 (11): 1045–53. doi:10.1007/s11745-004-1329-9. PMID 15726818.
White S, Zheng J, Zhang Y (2005). "The structural biology of type II fatty acid biosynthesis". Annu Rev Biochem. 74: 791–831. doi:10.1146/annurev.biochem.74.082803.133524. PMID 15952903.
Ohlrogge J, Jaworski J (1997). "Regulation of fatty acid synthesis". Annu Rev Plant Physiol Plant Mol Biol. 48: 109–136. doi:10.1146/annurev.arplant.48.1.109. PMID 15012259.
Dubey V, Bhalla R, Luthra R (2003). "An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants" (PDF). J Biosci. 28 (5): 637–46. doi:10.1007/BF02703339. PMID 14517367. Archived from the original (PDF) on 15 April 2007.
Kuzuyama T, Seto H (2003). "Diversity of the biosynthesis of the isoprene units". Nat Prod Rep. 20 (2): 171–83. doi:10.1039/b109860h. PMID 12735695.
Grochowski L, Xu H, White R (May 2006). "Methanocaldococcus jannaschii Uses a Modified Mevalonate Pathway for Biosynthesis of Isopentenyl Diphosphate". J Bacteriol. 188 (9): 3192–8. doi:10.1128/JB.188.9.3192-3198.2006. PMC 1447442. PMID 16621811.
Lichtenthaler H (1999). "The 1-Ddeoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants". Annu Rev Plant Physiol Plant Mol Biol. 50: 47–65. doi:10.1146/annurev.arplant.50.1.47. PMID 15012203.
Schroepfer G (1981). "Sterol biosynthesis". Annu Rev Biochem. 50: 585–621. doi:10.1146/annurev.bi.50.070181.003101. PMID 7023367.
Lees N, Skaggs B, Kirsch D, Bard M (1995). "Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisiae—a review". Lipids. 30 (3): 221–6. doi:10.1007/BF02537824. PMID 7791529.
Himmelreich R, Hilbert H, Plagens H, Pirkl E, Li BC, Herrmann R (November 1996). "Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae". Nucleic Acids Res. 24 (22): 4420–49. doi:10.1093/nar/24.22.4420. PMC 146264. PMID 8948633.
Guyton, Arthur C.; John E. Hall (2006). Textbook of Medical Physiology. Philadelphia: Elsevier. pp. 855–6. ISBN 0-7216-0240-1.
Ibba M, Söll D (2001). "The renaissance of aminoacyl-tRNA synthesis". EMBO Rep. 2 (5): 382–7. doi:10.1093/embo-reports/kve095. PMC 1083889. PMID 11375928. Archived from the original on 1 May 2011.
Lengyel P, Söll D (1969). "Mechanism of protein biosynthesis". Bacteriol Rev. 33 (2): 264–301. PMC 378322. PMID 4896351.
Rudolph F (1994). "The biochemistry and physiology of nucleotides". J Nutr. 124 (1 Suppl): 124S–127S. PMID 8283301. Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). "Pyrimidine and purine biosynthesis and degradation in plants". Annu Rev Plant Biol. 57: 805–36. doi:10.1146/annurev.arplant.57.032905.105421. PMID 16669783.
Stasolla C, Katahira R, Thorpe T, Ashihara H (2003). "Purine and pyrimidine nucleotide metabolism in higher plants". J Plant Physiol. 160 (11): 1271–95. doi:10.1078/0176-1617-01169. PMID 14658380.
Davies O, Mendes P, Smallbone K, Malys N (2012). "Characterisation of multiple substrate-specific (d)ITP/(d)XTPase and modelling of deaminated purine nucleotide metabolism". BMB Reports. 45 (4): 259–64. doi:10.5483/BMBRep.2012.45.4.259. PMID 22531138.
Smith J (1995). "Enzymes of nucleotide synthesis". Curr Opin Struct Biol. 5 (6): 752–7. doi:10.1016/0959-440X(95)80007-7. PMID 8749362.
Testa B, Krämer S (2006). "The biochemistry of drug metabolism—an introduction: part 1. Principles and overview". Chem Biodivers. 3 (10): 1053–101. doi:10.1002/cbdv.200690111. PMID 17193224.
Danielson P (2002). "The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans". Curr Drug Metab. 3 (6): 561–97. doi:10.2174/1389200023337054. PMID 12369887.
King C, Rios G, Green M, Tephly T (2000). "UDP-glucuronosyltransferases". Curr Drug Metab. 1 (2): 143–61. doi:10.2174/1389200003339171. PMID 11465080.
Sheehan D, Meade G, Foley V, Dowd C (November 2001). "Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily". Biochem J. 360 (Pt 1): 1–16. doi:10.1042/0264-6021:3600001. PMC 1222196. PMID 11695986.
Galvão T, Mohn W, de Lorenzo V (2005). "Exploring the microbial biodegradation and biotransformation gene pool". Trends Biotechnol. 23 (10): 497–506. doi:10.1016/j.tibtech.2005.08.002. PMID 16125262.
Janssen D, Dinkla I, Poelarends G, Terpstra P (2005). "Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities". Environ Microbiol. 7 (12): 1868–82. doi:10.1111/j.1462-2920.2005.00966.x. PMID 16309386.
Davies K (1995). "Oxidative stress: the paradox of aerobic life". Biochem Soc Symp. 61: 1–31. doi:10.1042/bss0610001. PMID 8660387.
Tu B, Weissman J (2004). "Oxidative protein folding in eukaryotes: mechanisms and consequences". J Cell Biol. 164 (3): 341–6. doi:10.1083/jcb.200311055. PMC 2172237. PMID 14757749.
Sies H (1997). "Oxidative stress: oxidants and antioxidants" (PDF). Exp Physiol. 82 (2): 291–5. doi:10.1113/expphysiol.1997.sp004024. PMID 9129943.
Vertuani S, Angusti A, Manfredini S (2004). "The antioxidants and pro-antioxidants network: an overview". Curr Pharm Des. 10 (14): 1677–94. doi:10.2174/1381612043384655. PMID 15134565.
von Stockar U, Liu J (1999). "Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth". Biochim Biophys Acta. 1412 (3): 191–211. doi:10.1016/S0005-2728(99)00065-1. PMID 10482783.
Demirel Y, Sandler S (2002). "Thermodynamics and bioenergetics". Biophys Chem. 97 (2–3): 87–111. doi:10.1016/S0301-4622(02)00069-8. PMID 12050002.
Albert R (2005). "Scale-free networks in cell biology". J Cell Sci. 118 (Pt 21): 4947–57. arXiv:q-bio/0510054. doi:10.1242/jcs.02714. PMID 16254242.
Brand M (1997). "Regulation analysis of energy metabolism". J Exp Biol. 200 (Pt 2): 193–202. PMID 9050227.
Soyer O, Salathé M, Bonhoeffer S (2006). "Signal transduction networks: topology, response and biochemical processes". J Theor Biol. 238 (2): 416–25. doi:10.1016/j.jtbi.2005.05.030. PMID 16045939.
Salter M, Knowles R, Pogson C (1994). "Metabolic control". Essays Biochem. 28: 1–12. PMID 7925313.
Westerhoff H, Groen A, Wanders R (1984). "Modern theories of metabolic control and their applications (review)". Biosci Rep. 4 (1): 1–22. doi:10.1007/BF01120819. PMID 6365197.
Fell D, Thomas S (1995). "Physiological control of metabolic flux: the requirement for multisite modulation". Biochem J. 311 (Pt 1): 35–9. PMC 1136115. PMID 7575476.
Hendrickson W (2005). "Transduction of biochemical signals across cell membranes". Q Rev Biophys. 38 (4): 321–30. doi:10.1017/S0033583506004136. PMID 16600054.
Cohen P (2000). "The regulation of protein function by multisite phosphorylation—a 25 year update". Trends Biochem Sci. 25 (12): 596–601. doi:10.1016/S0968-0004(00)01712-6. PMID 11116185.
Lienhard G, Slot J, James D, Mueckler M (1992). "How cells absorb glucose". Sci Am. 266 (1): 86–91. Bibcode:1992SciAm.266a..86L. doi:10.1038/scientificamerican0192-86. PMID 1734513.
Roach P (2002). "Glycogen and its metabolism". Curr Mol Med. 2 (2): 101–20. doi:10.2174/1566524024605761. PMID 11949930.
Newgard C, Brady M, O'Doherty R, Saltiel A (2000). "Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1" (PDF). Diabetes. 49 (12): 1967–77. doi:10.2337/diabetes.49.12.1967. PMID 11117996.
Romano A, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol. 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol. Advances in Microbial Physiology. 40: 353–99. doi:10.1016/S0065-2911(08)60135-6. ISBN 978-0-12-027740-7. PMID 9889982.
Ouzounis C, Kyrpides N (1996). "The emergence of major cellular processes in evolution". FEBS Lett. 390 (2): 119–23. doi:10.1016/0014-5793(96)00631-X. PMID 8706840.
Caetano-Anolles G, Kim HS, Mittenthal JE (2007). "The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture". Proc Natl Acad Sci USA. 104 (22): 9358–63. Bibcode:2007PNAS..104.9358C. doi:10.1073/pnas.0701214104. PMC 1890499. PMID 17517598.
Schmidt S, Sunyaev S, Bork P, Dandekar T (2003). "Metabolites: a helping hand for pathway evolution?". Trends Biochem Sci. 28 (6): 336–41. doi:10.1016/S0968-0004(03)00114-2. PMID 12826406.
Light S, Kraulis P (2004). "Network analysis of metabolic enzyme evolution in Escherichia coli". BMC Bioinformatics. 5: 15. doi:10.1186/1471-2105-5-15. PMC 394313. PMID 15113413. Alves R, Chaleil R, Sternberg M (2002). "Evolution of enzymes in metabolism: a network perspective". J Mol Biol. 320 (4): 751–70. doi:10.1016/S0022-2836(02)00546-6. PMID 12095253.
Kim HS, Mittenthal JE, Caetano-Anolles G (2006). "MANET: tracing evolution of protein architecture in metabolic networks". BMC Bioinformatics. 7: 351. doi:10.1186/1471-2105-7-351. PMC 1559654. PMID 16854231.
Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (2001). "Small-molecule metabolsim: an enzyme mosaic". Trends Biotechnol. 19 (12): 482–6. doi:10.1016/S0167-7799(01)01813-3. PMID 11711174.
Spirin V, Gelfand M, Mironov A, Mirny L (June 2006). "A metabolic network in the evolutionary context: Multiscale structure and modularity". Proc Natl Acad Sci USA. 103 (23): 8774–9. Bibcode:2006PNAS..103.8774S. doi:10.1073/pnas.0510258103. PMC 1482654. PMID 16731630.
Lawrence J (2005). "Common themes in the genome strategies of pathogens". Curr Opin Genet Dev. 15 (6): 584–8. doi:10.1016/j.gde.2005.09.007. PMID 16188434. Wernegreen J (2005). "For better or worse: genomic consequences of intracellular mutualism and parasitism". Curr Opin Genet Dev. 15 (6): 572–83. doi:10.1016/j.gde.2005.09.013. PMID 16230003.
Pál C, Papp B, Lercher M, Csermely P, Oliver S, Hurst L (2006). "Chance and necessity in the evolution of minimal metabolic networks". Nature. 440 (7084): 667–70. Bibcode:2006Natur.440..667P. doi:10.1038/nature04568. PMID 16572170.
Rennie M (1999). "An introduction to the use of tracers in nutrition and metabolism". Proc Nutr Soc. 58 (4): 935–44. doi:10.1017/S002966519900124X. PMID 10817161.
Phair R (1997). "Development of kinetic models in the nonlinear world of molecular cell biology". Metabolism. 46 (12): 1489–95. doi:10.1016/S0026-0495(97)90154-2. PMID 9439549.
Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (2007). "How many genes are there in plants (... and why are they there)?". Curr Opin Plant Biol. 10 (2): 199–203. doi:10.1016/j.pbi.2007.01.004. PMID 17289424.
Borodina I, Nielsen J (2005). "From genomes to in silico cells via metabolic networks". Curr Opin Biotechnol. 16 (3): 350–5. doi:10.1016/j.copbio.2005.04.008. PMID 15961036.
Gianchandani E, Brautigan D, Papin J (2006). "Systems analyses characterize integrated functions of biochemical networks". Trends Biochem Sci. 31 (5): 284–91. doi:10.1016/j.tibs.2006.03.007. PMID 16616498.
Duarte NC, Becker SA, Jamshidi N, et al. (February 2007). "Global reconstruction of the human metabolic network based on genomic and bibliomic data". Proc. Natl. Acad. Sci. U.S.A. 104 (6): 1777–82. Bibcode:2007PNAS..104.1777D. doi:10.1073/pnas.0610772104. PMC 1794290. PMID 17267599.
Goh KI, Cusick ME, Valle D, Childs B, Vidal M, Barabási AL (May 2007). "The human disease network". Proc. Natl. Acad. Sci. U.S.A. 104 (21): 8685–90. Bibcode:2007PNAS..104.8685G. doi:10.1073/pnas.0701361104. PMC 1885563. PMID 17502601.
Lee DS, Park J, Kay KA, Christakis NA, Oltvai ZN, Barabási AL (July 2008). "The implications of human metabolic network topology for disease comorbidity". Proc. Natl. Acad. Sci. U.S.A. 105 (29): 9880–9885. Bibcode:2008PNAS..105.9880L. doi:10.1073/pnas.0802208105. PMC 2481357. PMID 18599447.
Csete M, Doyle J (2004). "Bow ties, metabolism and disease". Trends Biotechnol. 22 (9): 446–50. doi:10.1016/j.tibtech.2004.07.007. PMID 15331224.
Ma HW, Zeng AP (2003). "The connectivity structure, giant strong component and centrality of metabolic networks". Bioinformatics. 19 (11): 1423–30. CiteSeerX 10.1.1.605.8964. doi:10.1093/bioinformatics/btg177. PMID 12874056.
Zhao J, Yu H, Luo JH, Cao ZW, Li YX (2006). "Hierarchical modularity of nested bow-ties in metabolic networks". BMC Bioinformatics. 7: 386. doi:10.1186/1471-2105-7-386. PMC 1560398. PMID 16916470.
Thykaer J, Nielsen J (2003). "Metabolic engineering of beta-lactam production". Metab Eng. 5 (1): 56–69. doi:10.1016/S1096-7176(03)00003-X. PMID 12749845. González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade J, Vasconcelos I, Soucaille P (2005). "Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol". Metab Eng. 7 (5–6): 329–36. doi:10.1016/j.ymben.2005.06.001. PMID 16095939. Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, Wubbolts M, Raeven L (2003). "Metabolic engineering for microbial production of shikimic acid". Metab Eng. 5 (4): 277–83. doi:10.1016/j.ymben.2003.09.001. PMID 14642355.
Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). "Metabolic engineering". Annu Rev Biomed Eng. 1: 535–57. doi:10.1146/annurev.bioeng.1.1.535. PMID 11701499.
"Metabolism". The Online Etymology Dictionary. Retrieved 20 February 2007.
Leroi, Armand Marie (2014). The Lagoon: How Aristotle Invented Science. Bloomsbury. pp. 400–401. ISBN 978-1-4088-3622-4.
Dr. Abu Shadi Al-Roubi (1982), "Ibn Al-Nafis as a philosopher", Symposium on Ibn al-Nafis, Second International Conference on Islamic Medicine: Islamic Medical Organization, Kuwait (cf. Ibn al-Nafis As a Philosopher, Encyclopedia of Islamic World [1])
Eknoyan G (1999). "Santorio Sanctorius (1561–1636) – founding father of metabolic balance studies". Am J Nephrol. 19 (2): 226–33. doi:10.1159/000013455. PMID 10213823.
Williams, H. S. (1904) A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) Retrieved on 26 March 2007
Dubos J. (1951). "Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind". Trends Biotechnol. 13 (12): 511–515. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
Kinne-Saffran E, Kinne R (1999). "Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs". Am J Nephrol. 19 (2): 290–4. doi:10.1159/000013463. PMID 10213830.
Eduard Buchner's 1907 Nobel lecture at http://nobelprize.org Accessed 20 March 2007
Kornberg H (2000). "Krebs and his trinity of cycles". Nat Rev Mol Cell Biol. 1 (3): 225–8. doi:10.1038/35043073. PMID 11252898.
Krebs HA, Henseleit K (1932). "Untersuchungen über die Harnstoffbildung im tierkorper". Z. Physiol. Chem. 210: 33–66. doi:10.1515/bchm2.1932.210.1-2.33.
Krebs H, Johnson W (April 1937). "Metabolism of ketonic acids in animal tissues". Biochem J. 31 (4): 645–60. doi:10.1042/bj0310645. PMC 1266984. PMID 16746382. |